X-MOL: 【纳米】DNA支架指导的病毒蛋白多级有序组装及可编程动态调控
核酸和蛋白质是生命体基础结构与功能单位的最重要成分,同时也是优秀的分子自组装纳米材料。如果我们充分了解核酸和蛋白质的精细结构与功能之间的联系,在此基础上实现两种组分组装驱动力的精准操控,就可能进行人工设计和建造新型超分子材料和生物纳米机器。蛋白质作为生命活动的主要承担者,具有结构多样、功能丰富的特点。但是,氨基酸组合的多样性和折叠机制的复杂性,使得多组分、各向异性、多级组装过程中蛋白模块的相对取向、化学计量数以及反应节点难以控制。如何对组装过程进行控制,实现程序性动态调控,构筑更高层次的杂合结构,是生物材料可控组装研究及应用面临的一个主要挑战。DNA的组装模式则相对简单,可操作性更高。近年来,DNA纳米技术取得众多令人瞩目的研究成果。研究人员在计算机帮助下构建了一系列精致的DNA多维结构,利用其伸出的DNA捕获链杂交蛋白表面偶联的DNA片段,可指导蛋白客体在预定位置有序排布。然而,利用蛋白质表面氨基酸残基进行DNA共价交联的常规方法,有着一些难以规避的缺点,包括修饰基团对功能蛋白活性的损害,修饰点与取向的不可控性以及操作体系不适于体内自组装过程等,限制了DNA-蛋白质复合纳米结构的可编程构筑。
针对这一挑战,中科院苏州纳米所王强斌研究员课题组与美国Emory大学柯勇刚教授合作,探索DNA纳米技术结合病毒纳米颗粒的增殖装配机制来制造更加复杂可控的核酸-蛋白质新型组装系统。病毒是自然界进化发展的一种典型的自组体。它的装配过程具有很高的特异性和效率,能够在较短时间内利用弱键协同作用组装生产出大量的病毒颗粒。目前已经知道,病毒基因组与衣壳蛋白的互作规律对整个病毒装配过程起到非常重要的作用。假设人们能在纳米尺度下对病毒基因组相关序列的状态进行精确操纵,则有可能实现人为调控病毒蛋白的各个复合组装步骤。
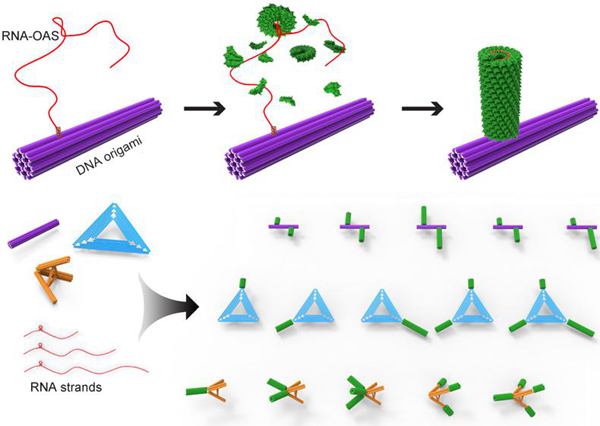
图1、利用病毒蛋白与基因组RNA内在作用机制在DNA支架上进行原位组装体系的设计,展示DNA-蛋白复合结构的可控构筑。
首先他们以烟草花叶病毒(tobacco mosaic virus, TMV)作为模式系统,探索了不同条件下TMV基因组RNA与衣壳蛋白的互作规律及其对病毒颗粒的原位装配调控(图1)。TMV基因组特定的组装起始序列可有效引导核酸与病毒衣壳蛋白的特异性结合并引发体外重构组装,并且病毒蛋白管的组装长度是由RNA长度决定的,从而为蛋白域的精确调控提供了可能。研究人员构建了一维到三维DNA origami模板作为支架结合不同长度的TMV RNA重组序列,引导后续的原位组装过程。通过支架表面结合位点和序列的设计,不仅能实现TMV病毒蛋白管在DNA支架特定位置按一定组装程序进行定向装配生长,还完成蛋白管原位组装长度的有效调控。这项工作发表在《美国化学会志》杂志上 (J. Am. Chem. Soc. 2018, 140, 26, 8074)。

图2、基于DNA origami支架提供的锚定平台实现核酸-蛋白质多级有序组装及可编程动态调控示意图。
在此基础上,他们进一步对病毒基因组相关RNA序列的活性状态进行了巧妙设计,实现病毒蛋白组装步骤的动态调控(图2)。首先,通过DNA origami上伸出的互补交联序列的设计,进行RNA长链的捕获研究,实现RNA的有序排布;然后,RNA长链上伸出的病毒组装起始引导序列结合TMV病毒衣壳蛋白亚基,启动持续组装直至到达DNA origami支架表面而暂停;最后,通过控制DNA origami支架表面固定链的有序打开,逐步释放预定长度的RNA基序,不仅可以实现蛋白组装程序的精确控制,还能操纵组装体按预定路线进行实时精确移动。利用DNA支架提供的锚定平台综合病毒蛋白装配机制,他们设计构建的核酸-蛋白质动态组装系统在以下几方面具有重要意义。1,首次提出利用DNA支架预铺设开关路径策略,通过DNA origami上简单的锚定序列将RNA长链进行可控化编程,成功将病毒蛋白装配持续反应进程中难以实现的调控点进行转化,实现精细分区段控制。2,有望实现不同病毒蛋白亚基突变体的分区段精确掺入,可用于制备具有多种功能化区域的病毒样组装体。3,与已有的核酸-蛋白质多层级组装体构建方法相比,此课题不需要对蛋白基元进行特殊处理,结合分子探针,有望用于细胞内病毒组装体系的实时装配检测。该成果近期发表在《美国化学会志》杂志上并入选JACS亮点论文。
Programming Dynamic Assembly of Viral Proteins with DNA Origami. Kun Zhou, Yihao Zhou, Victor Pan, Qiangbin Wang, Yonggang Ke. J. Am. Chem. Soc., 2020, 142, 5929-5932, DOI: 10.1021/jacs.9b13773.
Spotlights on Recent JACS Publications. J. Am. Chem. Soc., 2020, 142, 6835-6836, DOI: 10.1021/jacs.0c03750.
https://pubs.acs.org/doi/abs/10.1021/jacs.0c03750.
附件下载:
